Інсерцыя (генетыка)
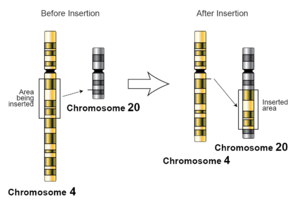
У генетыцы інсерцыя — гэта даданне адной або некалькіх пар асноў нуклеатыдаў у паслядоўнасць ДНК. Гэта часта можа адбывацца ў мікрасатэлітных рэгіёнах з-за памылкі ДНК-палімеразы пры рэплікацыі. Устаўкі могуць быць любога памеру: ад адной пары асноў, няправільна ўстаўленай у паслядоўнасць ДНК, да ўчастка адной храмасомы, устаўленай у іншую. Мяркуецца, што механізм самых маленькіх мутацый інсерцыі адзінай асновы заключаецца ў падзеле пары асноў паміж матрычным і праймерным ланцужкамі з наступным нагрувашчваннем несуседніх асноў, якое можа адбывацца лакальна ў актыўным цэнтры ДНК-палімеразы.[1] На храмасомным узроўні пад інсерцыяй маецца на ўвазе ўстаўка паслядоўнасці ў храмасому з іншай храмасомы. Гэта можа адбыцца з-за неаднолькавага красінговеру падчас меёзу.
Далучэнне N-вобласці — гэта далучэнне канцавой дэзаксінуклеатыдылтрансферазай некадуючых нуклеатыдаў падчас рэкамбінацыі.
Інсерцыя P нуклеатыдаў — гэта ўстаўка паліндромных паслядоўнасцей, закадаваных канцамі рэкамбінуючых сегментаў гена.
Трынуклеатыдныя паўторы класіфікуюцца як інсерцыйныя мутацыі[2][3], а часам і як асобны клас мутацый.[4]
Метады правіць
Нуклеаза цынкавага пальца (ZFN), эфектарная нуклеаза, падобная на актыватар транскрыпцыі (TALEN) і рэдагаванне гена CRISPR — гэта тры асноўныя метады, якія выкарыстоўваліся ў папярэдніх даследаваннях для дасягнення ўстаўкі гена. Інструменты CRISPR/Cas ужо сталі аднымі з найбольш часта выкарыстоўваемых метадаў сучасных даследаванняў.
На аснове інструментаў CRISPR/Cas ужо былі распрацаваны розныя сістэмы для дасягнення пэўных функцый. Напрыклад, адной з стратэгій з’яўляецца сістэма двухланцуговых нуклеаз з выкарыстаннем бялку Cas9 з адной кіруючай РНК (sgRNA), а затым дасягненне інсерцыі гена шляхам злучэння канцоў або дзялення клетак з дапамогай сістэмы рэпарацыі ДНК.[5] Іншым прыкладам з’яўляецца сістэма прайм-рэдагавання, якая выкарыстоўвае ніказу Cas9 і кіруючую прайм-РНК для рэдагавання (pegRNA), якая нясе таргетныя гены.[5]
Адным з абмежаванняў сучаснай тэхналогіі з’яўляецца тое, што памер для дакладнай устаўкі ДНК недастаткова вялікі[6], каб задаволіць попыт пры даследаваннях геному. Транспазіцыя ДНК з напраўляючай РНК — гэта новая вобласць для вырашэння гэтай праблемы.[7] Больш эфектыўныя метады плануецца распрацаваць і прымяніць у галіне геномнай інжынерыі.
Эфекты правіць
Устаўкі могуць быць асабліва небяспечнымі, калі яны адбываюцца ў экзоне, вобласці кадавання амінакіслот гена. Мутацыя зруху рамкі счытвання, якая змяняе рамку счытвання гена, узнікае, калі колькасць устаўленых нуклеатыдаў не дзеліцца на тры, то-бок колькасці нуклеатыдаў на кадон. Мутацыі зруху рамкі счытвання змяняюць усе амінакіслоты, закадаваныя генам пасля мутацыі. Звычайна ўстаўкі і наступная мутацыя зруху рамкі счытвання прыводзяць да таго, што актыўная трансляцыя гена сутыкаецца з заўчасным стоп-кадонам, што прыводзіць да спынення трансляцыі і прадукцыі ўсечанага бялку. Транскрыпты, якія нясуць мутацыю зруху рамкі счытвання, таксама могуць пагаршацца шляхам нонсэнс-апасродкаванага распаду падчас трансляцыі, што не прыводзіць да ўзнікнення бялковага прадукту. У выпадку трансляцыі, усечаныя бялкі часта не могуць функцыянаваць належным чынам і могуць прывесці да любой колькасці генетычных парушэнняў у залежнасці ад гена, у якім адбываецца інсерцыя.[8]
Інсерцыі ў рамку счытвання адбываюцца, калі рамка счытвання не змяняецца ў выніку ўстаўкі; колькасць устаўленых нуклеатыдаў дзеліцца на тры. Рамка счытвання застаецца непашкоджанай пасля ўстаўкі, і трансляцыя, хутчэй за ўсё, завершыцца, калі ўстаўленыя нуклеатыды не кадуюць стоп-кадон. Аднак з-за ўстаўленых нуклеатыдаў гатовы бялок будзе мець, у залежнасці ад памеру ўстаўкі, некалькі новых амінакіслот, якія могуць паўплываць на функцыю бялку.
Гл. таксама правіць
Крыніцы правіць
- ↑ Banavali, Nilesh K. (2013). "Partial Base Flipping is Sufficient for Strand Slippage near DNA Duplex Termini". Journal of the American Chemical Society. 135 (22): 8274–8282. doi:10.1021/ja401573j. PMID 23692220.
- ↑ Mechanisms: Genetic Variation: Types of Mutations(недаступная спасылка). Evolution 101: Understanding Evolution For Teachers. University of California Museum of Paleontology. Архівавана з першакрыніцы 14 красавіка 2009. Праверана 19 верасня 2009. ] Understanding Evolution For Teachers Home. Retrieved on September 19, 2009
- ↑ Brown, Terence A. (2007). "16 Mutations and DNA Repair". Genomes 3. Garland Science. p. 510. ISBN 978-0-8153-4138-3.
- ↑ Faraone, Stephen V.; Tsuang, Ming T.; Tsuang, Debby W. (1999). "5 Molecular Genetics and Mental Illness: The Search for Disease Mechanisms: Types of Mutations". Genetics of Mental Disorders: A Guide for Students, Clinicians, and Researchers. Guilford Press. p. 145. ISBN 978-1-57230-479-6.
- ↑ а б Anzalone, Andrew V.; Koblan, Luke W.; Liu, David R. (2020). "Genome editing with CRISPR–Cas nucleases, base editors, transposases and prime editors". Nature Biotechnology. 38 (7): 824–844. doi:10.1038/s41587-020-0561-9. PMID 32572269. S2CID 256820370.
{{cite journal}}: Праверце значэнне|s2cid=(даведка) - ↑ Sun, Chao; Lei, Yuan; Li, Boshu; Gao, Qiang; Li, Yunjia; Cao, Wen; Yang, Chao; Li, Hongchao; Wang, Zhiwei; Li, Yan; Wang, Yanpeng; Liu, Jun; Zhao, Kevin Tianmeng; Gao, Caixia (2023). "Precise integration of large DNA sequences in plant genomes using PrimeRoot editors". Nature Biotechnology: 1–12. doi:10.1038/s41587-023-01769-w. PMID 37095350. S2CID 258311438.
{{cite journal}}: Праверце значэнне|s2cid=(даведка) - ↑ Wang, Joy Y.; Doudna, Jennifer A. (2023). "CRISPR technology: A decade of genome editing is only the beginning". Science. 379 (6629): eadd8643. doi:10.1126/science.add8643. PMID 36656942. S2CID 255966509.
- ↑ Shmilovici, A.; Ben-Gal, I. (2007). "Using a VOM Model for Reconstructing Potential Coding Regions in EST Sequences" (PDF). Journal of Computational Statistics. 22 (1): 49–69. doi:10.1007/s00180-007-0021-8. S2CID 2737235.
Дадатковая літаратура правіць
- Pierce, Benjamin A. (2013). Genetics: A Conceptual Approach (5th ed.). W. H. Freeman. ISBN 978-1-4641-5084-5.